|
鹿児島県肉用牛改良研究所新技術開発研究室、東京大学大学院新領域創成科学研究科、畜産技術協会、琉球大学農学部らの共同研究グループは、黒毛和種の胎子、子牛、成牛の主要31臓器、合計124サンプルを用い、詳細な大規模遺伝子発現解析を行いました。 |
<発表概要>
(研究の背景)
黒毛和種は、筋肉内に脂肪が綺麗に蓄積する脂肪交雑、いわゆる「霜降り」が特徴で、おいしい牛肉を生産する我が国が誇る肉用牛です(図1、用語解説1)。国内で170万頭飼育されており、子牛のほとんどが人工授精で生産されています。人工授精には、遺伝的に優れた種雄牛の凍結精液が利用されるため、優秀な種雄牛からは数万頭から数十万頭もの子牛が生まれています。一方で、優秀な種雄牛の産子が増えることで、近交 の度合いが高まり(用語説明2)、胎子や子牛の死亡など遺伝性疾患の発生リスクが増すことになります。
このような背景から、黒毛和種の持続的生産、改良のため、遺伝性疾患の発生によって生じる損失を防ぐことを目的とし、共同研究グループでは種雄牛を造成している機関と協力して、遺伝性疾患の原因となる変異を迅速に見つけるための「和牛ゲノムデータベース」の構築を続けてきました(用語解説3)。
今回、変異を有する遺伝子の機能解析を促進するため、2種類の次世代シーケンサーを使用し、黒毛和種の胎子、子牛、成牛の主要31臓器、合計124サンプルを用いて大規模遺伝子発現解析を行い、その情報を和牛ゲノムデータベースに格納しました(図2)。このような黒毛和種の高精度な大規模遺伝子発現解析は初めての取組で、今後、黒毛和種の遺伝性疾患に加え、経済形質に関わる遺伝子の機能解明を促進する基礎データになることが期待されます。
(研究の成果)
黒毛和種の胎子(30日齢、60日齢、90日齢)、子牛(生後10日、生後23日)、成牛の主要31臓器、合計124サンプルからRNAを抽出し、短鎖と長鎖の配列を解析できる2種の次世代シーケンサーを併用し解読を行った後(用語説明4)、遺伝子発現の臓器特異性や時期特異性について解析を行いました。また、長鎖の次世代シーケンサーを用いることで、これまで報告されていない232個のアイソフォームも検出しました(用語説明5)。これらの解析データは、和牛ゲノムデータベース(図2)や日本DNAデータバンク(DDBJ)から利用可能です。
本研究は、日本中央競馬会畜産振興事業の支援で行われました。
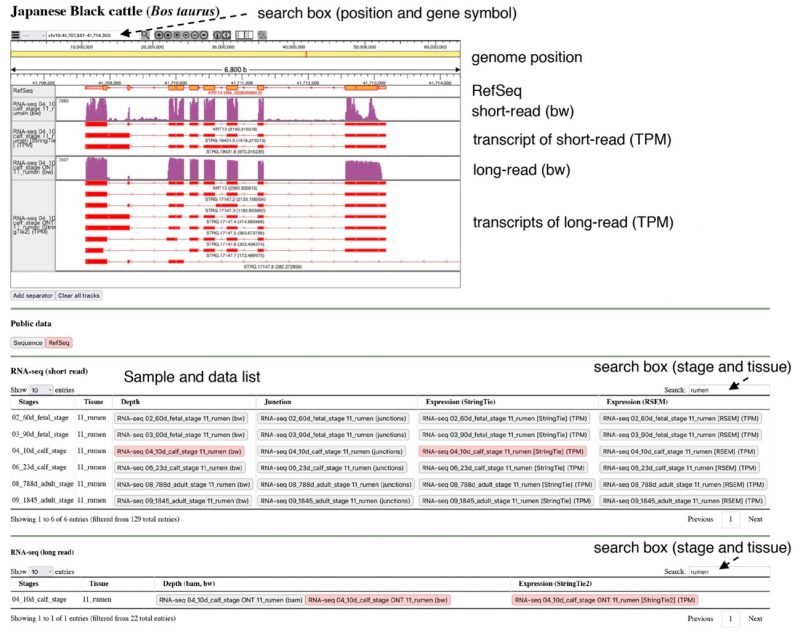
図2)和牛ゲノムデータベース(https://wagyu.hgc.jp)。web上で利用可能である。
<用語解説>
1)黒毛和種:和牛は、黒毛和種、褐毛和種、日本短角種、無角和種の4品種で構成され、黒毛和種は、その95%を占め、肉質、特に脂肪交雑(霜降り)で優れた品種です。
2)近交の度合い:近親交配の度合い。専門的には、個体が持つ2つの相同する染色体領域が共通先祖由来である確率、近交係数として表現されます。
3)和牛ゲノムデータベース:黒毛和種の重要な種雄牛を次世代シーケンサーで解読し、変異を格納したデータベースで、和牛ゲノムデータベース協議会(代表機関 公益社団法人畜産技術協会)が管理しています。今回の成果は、遺伝子発現情報を、和牛ゲノムデータベース(図2)に追加したもので、日本DNAデータバンク(DDBJ)からも利用可能です。
4)短鎖と長鎖の配列を解析できる次世代シーケンサー:これまで、次世代シーケンサーでは、塩基の配列情報を解読するため、短鎖の塩基断片(100から300塩基程度)に切断して解析しており、塩基の読み取りは正確ではあるが、解読された短鎖の塩基断片から元の遺伝子構造を再構築する必要がありました。一方、最近登場した長鎖の塩基断片(1万塩基以上)の配列を解析できる次世代シーケンサーは、塩基の読み取りの正確さは短鎖の次世代シーケンサーに劣るものの、遺伝子構造をそのまま解析できる利点があります。今回の解析では、2種の次世代シーケンサーを併用し解読することで、遺伝子発現と遺伝子構造の詳細な解析が可能になりました。
5)アイソフォーム :染色体上で遺伝子は、蛋白質の情報を含むエクソンが、蛋白質の情報を含まないイントロンで分断された状態で存在しています。遺伝子から蛋白質が合成される中間段階で作られる前駆体のmRNAは、イントロンも含む形ですが、成熟型のmRNAではスプライシングという過程を経て、イントロンが取り除かれ、エクソンが結合し、蛋白質を合成する鋳型が形成されます。スプライシングの過程で、エクソンの結合やイントロンの取り除かれ方が変化することがあり、1つの遺伝子から複数のmRNAが作成され、結果としてアイソフォームと呼ばれる複数の蛋白質が合成されることになります。今回の解析では、これまで報告されていない232個のアイソフォーム も検出しました。
<論文情報>
(1) タイトル;Comprehensive analysis of 124 transcriptomes from 31 tissues in developing, juvenile, and adult Japanese Black cattle (黒毛和種における胎子、子牛、成牛の主要31臓器、合計124サンプルの遺伝子発現の総合カタログ)
(2) 雑誌名:DNA Research
(3) 著者:Taichi Arishima1*, Hiroyuki Wakaguri2*, Ryotaro Nakashima1, Seigo Sakakihara1, Keisuke Kawashima1, Yoshikazu Sugimoto3, Yutaka Suzuki2+, and Shinji Sasaki4+(1鹿児島県肉用牛改良研究所新技術開発研究室、2東京大学大学院新領域創成科学研究科、3畜産技術協会、4琉球大学農学部、*筆頭著者、+責任著者)
(4) DOI番号: 10.1093/dnares/dsac032
(5) アブストラクトURL: https://doi.org/10.1093/dnares/dsac032


