|
琉球大学大学院医学研究科の松波雅俊助教、今村美菜子准教授、木村亮介教授、前田士郎教授、理化学研究所生命医科学研究センターの劉暁渓客員研究員(静岡県立総合病院研究員)、寺尾知可史チームリーダー(静岡県立総合病院 免疫研究部長、静岡県立大学 特任教授)、国立長寿医療研究センターの尾崎浩一部長らの研究チームによる研究成果が、進化生物学の学術雑誌「Molecular Biology and Evolution」誌に掲載されました。 |
<発表のポイント>
-
内閣府、文部科学省、沖縄県の支援を受けた沖縄バイオインフォメーションバンク、および国立長寿医療研究センターバイオバンクの日本人協力者20,366名のゲノム解析を実施した。
-
集団遺伝解析の結果、琉球列島の各島嶼(沖縄本島、宮古諸島、八重山諸島、慶良間諸島・久米島)の遺伝的背景は、わずかではあるがそれぞれ異なることが明らかになった。
-
ヒトゲノム全域にわたる解析により、琉球列島集団および本土集団の自然選択を反映する複数のゲノム領域を発見した。その中には、両集団に共通のものとそれぞれに特有のものがあり、両者の違いと共通性が示された。
-
6番染色体に位置する免疫に関連する領域(MHC領域)では琉球列島集団および本土集団で異なる対立遺伝子が選択されていた。
-
本研究の成果は、琉球列島および本土集団の遺伝的背景形成過程の理解の一助となるとともに、疾病構造の解明や遺伝的背景を考慮した健康増進に貢献することが期待される。
<発表概要>
① 研究の背景
現生人類(ホモ・サピエンス)は約10万年前にアフリカを旅立ってから、さまざまな環境に適応して生存してきました。このような環境への適応の歴史は、自然選択の痕跡としてヒトゲノム(注1)上に刻まれています(注2)。多様な人類集団のヒトゲノムを比較したこれまでの研究から、このようなゲノム領域は、高地環境への適応、食生活の変化や新たな病原体への耐性など、さまざまな環境要因と関係することが知られています(注3)。したがって、ゲノム上の自然選択領域を知ることは、それぞれの人類集団史の解明に繋がるとともに、地域における疾病構造の違いを理解する上でも重要と言えます。
日本人集団については、バイオバンク・ジャパン(注4)により収集された約17万人のヒトゲノム情報を、ASMC(注5)という遺伝統計解析手法を用いて解析し、それまでに同定されていた(注6)ADH1B、MHC、ALDH2を含む29か所の自然選択を受けたゲノム領域が報告されています(注7)。しかし、この報告では、日本人は単一の遺伝的背景を持つ集団として解析されていました。
過去の報告により、琉球列島と本土の人々との間には僅かながらも遺伝学的に違いがあることが明らかになっています(注8-9)。また、琉球大学を中心に行われた一連の研究から、琉球列島内においても、島嶼(とうしょ)ごとに遺伝的背景の多様性が示されています(注10-12)。しかし、琉球列島の各地域間の詳細な遺伝的背景の比較や、本土と琉球列島の遺伝的背景の違いを考慮した全ゲノムを網羅した大規模な自然選択の解析はこれまでに行われていませんでした。
② 研究内容
琉球大学と理化学研究所の共同研究チームは、沖縄バイオインフォメーションバンク (OBi)(注13)および国立長寿医療研究センターバイオバンク (NCGG)(注14)の研究協力者20,366名(OBi:6,613名, NCGG:13,753名)のヒトゲノム情報を解析しました。はじめに、機械学習を活用した集団遺伝学的解析により地域による遺伝的背景の違いを詳細に調べました。次に、地域による遺伝的背景の違いを考慮に入れたゲノム上の自然選択を受けた領域の検出を試みました。
琉球列島の集団遺伝構造:研究協力者20,366名のゲノム情報を用いて主成分分析 (PCA)(注15)を行った結果、先行研究と同様に琉球列島と本土は独立の集団を形成することが確認され、さらに琉球列島内においても地域によって遺伝的背景の違いがあると推察されました(図1B)。琉球列島内のさらに詳細な集団遺伝構造を推定するためにPCAと機械学習による次元削減法の一つであるUMAP(注16)を組み合わせたPCA-UMAPによる解析を実施しました。その結果、従来、琉球クラスターと呼ばれていた沖縄本島に加え、宮古諸島、八重山諸島、慶良間諸島・久米島はそれぞれ独立の集団を形成することがわかりました(図1C)。以上のことから、琉球列島内にも島嶼ごとに多様性が存在すると考えられました。
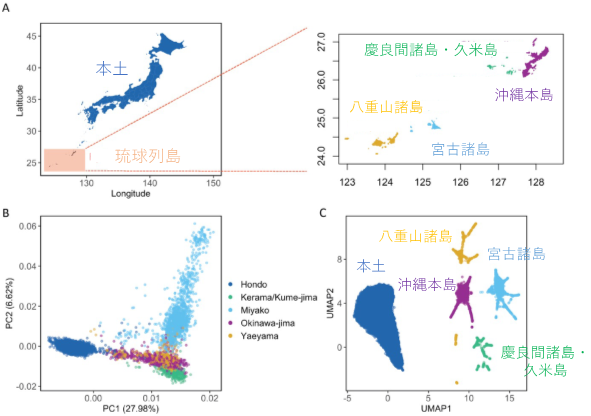
図1. 本土・琉球列島の集団遺伝構造 (A)本土と琉球列島および列島内の各島嶼の位置(B)PCAと(C)PCA-UMAPの結果 一つ一つの点が個人を表しており、点と点との距離は遺伝学的な違いを反映している。近い点ほど遺伝学的には近縁である。この図では出身地を元にした遺伝的背景の違いにより色分けをして表示している。
ゲノム上の自然選択の痕跡:つぎに、本土・琉球列島それぞれの集団についてゲノム上の自然選択の痕跡を探索しました。検出には、ゲノムのハプロタイプ構造(注17)を考慮して、比較的最近の自然選択まで推定可能なiHS(注18)とFastSMC(注19)という二つの手法を用いました。その結果(図2-3)、これまでに報告されている自然選択を受けたゲノム領域のうち、ADH clusterやEDAR, MHCは、両集団において検出されましたが、アルコール代謝に関係するALDH2領域は本土集団でのみ検出されました。
さらに、これまでに報告されていない3領域(IKZF2、HOXD、JMJD1C/CTNNA3)(注20-22)の自然選択の痕跡が琉球列島集団で検出されました。以上の結果から、両集団はある程度過去の集団史を共有していますが、それぞれの集団で異なる環境要因による独自の自然選択が働いたことが示唆されました。
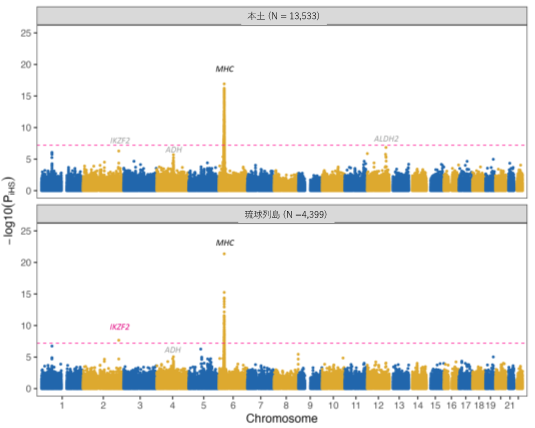
図2. iHSによる自然選択検出の結果:赤文字は新規に検出されたゲノム領域、灰色は統計的に有意ではないがピークが観察された領域。グラフのX軸は染色体上の位置、Y軸は自然選択の痕跡の強さを表す。プロットは各領域の解析結果を示し、Y軸の値が大きいほど強く自然選択の影響を受けたことを意味する。
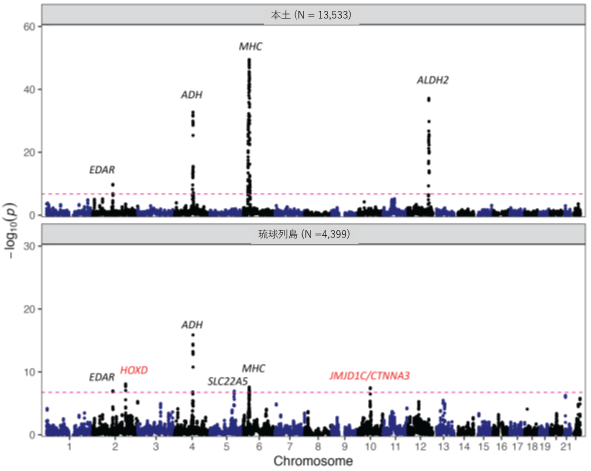
図3. FastSMCによる自然選択検出の結果:赤文字は新規に検出されたゲノム領域。グラフのX軸は染色体上の位置、Y軸は自然選択の痕跡の強さを表す。プロットは各領域の解析結果を示し、Y軸の値が大きいほど強く自然選択の影響を受けたことを意味する。
本土と琉球列島間でのHLA対立遺伝子の差:免疫に関係するMHC(HLA)領域(注23)は、本土と琉球列島の両方で自然選択の痕跡が検出されましたが、本土では、HLA-A*33:03-C*14:03-B*44:03-DRB1*13:02-DQB1*06:04-DPB1*04:01、琉球列島では、HLA-DRB1*15:01- DQB1*06:02と異なる対立遺伝子(注24)が最も強い選択を受けていました (表1)。琉球列島と本土で異なる対立遺伝子が選択されていることについては、どのような環境要因が関与しているのか、その詳細は未だ明らかではありません。これまでに、琉球列島で選択されているHLA-DRB1*15:01- DQB1*06:02を保有すると、1型糖尿病やHTLV-1関連脊髄症(HAM/TSP)(注25)の発症頻度は低く、ナルコプレシー(注26)の頻度は増すことが報告されています。琉球列島の人々の遺伝的背景の形成過程にこのような疾患が関与していたのかは現時点では明らかではなく、今後詳細に検討する必要があります。
表1. 本土と琉球列島間でのHLAハプロタイプ頻度の差
| HLAハプロタイプ | 頻度(%) | |
| 琉球列島集団 | 本土集団 | |
| HLA-A*33:03-C*14:03-B*44:03-DRB1*13:02-DQB1*06:04-DPB1*04:01 | 1.8 | 8.3 |
| HLA-DRB1*15:01- DQB1*06:02 | 19 | 9.3 |
③ 社会的意義・今後の予定
本研究では、全ゲノムの視点からみると本土と琉球列島に起こった自然選択は概ね同様ですが、一部異なることが示されました。今後は、なぜこのような違いが存在するのかを、それぞれの集団がどのような環境変化にさらされたかを調べることなどにより検討する必要があります。今回は琉球列島の各島嶼特有の自然選択の痕跡は検出されませんでしたが、島嶼ごとのさらに詳細な解析を実施するためには、より多くの協力者のゲノム検体を用いた解析が必要です。
今回、琉球列島および本土集団において自然選択の影響を受けたゲノム領域を検出することが出来ました。このようなゲノム上の自然選択領域を明らかにすることは、それぞれの人類集団がたどった環境への適応の歴史のみならず、地域における疾病構造の理解にもつながる可能性があります。各地域に特徴的なゲノム情報は、今後、各個人に適切な医療を提供する際にも重要な情報となります。現在、我々は沖縄バイオインフォメーションバンクで得られた臨床情報とゲノム情報の統合解析により、沖縄県民に最適な医療の構築を目指しています。
<用語解説>
(注1)ゲノム:ゲノムとは一つの生命体を形成し維持するのに必要な情報であり、別名生命の設計図とも呼ばれる。ヒトのゲノムは細胞の中に含まれるデオキシリボ核酸(DNA)のことであり、細胞の核にある染色体に主に含まれており、30〜32億文字からなる。この情報すべてをヒトゲノムと呼ぶ。
(注2)ゲノム上の自然選択:ゲノム上の対立遺伝子頻度(注24)は、生存に有利でも不利でもないときは無作為に変化するが、特定の環境で生存に有利な対立遺伝子が存在するとその頻度は増加すると考えられており、これを自然選択という。このようにして、過去の自然選択の痕跡がゲノム上に存在していると推察されている。
(注3)自然選択と疾患との関連を示す例:アフリカのマラリア高頻発地域の人類集団では、鎌状赤血球症の原因となるヘモグロビンβ鎖遺伝子の対立遺伝子の頻度が高くなっている。この対立遺伝子をもつと赤血球が自然に壊れる溶血性貧血を起こし、通常は生存には不利となる。ところが、マラリア原虫は赤血球に寄生することからマラリア感染機会の多い集団ではマラリア発症抑制に働き生存に有利となり選択された結果、この対立遺伝子頻度が他の集団より多くなっていると推察される。
(注4)バイオバンク・ジャパン:糖尿病やがんなど様々な疾患を持つ日本人約27万人を対象とした生体試料のバイオバンクで、東京大学医科学研究所内に設置されている。オーダーメイド医療の実現プログラム、ゲノム研究バイオバンク事業などを通じて、ゲノムDNAや血清サンプルを臨床情報とともに収集し、研究者へのデータ提供や分譲を行っている。
(注5)ASMC(the ascertained sequentially Markovian coalescent): ゲノム全領域における自然選択の痕跡を定量化する解析手法の一つ。各ゲノム領域内に含まれる複数のバリアントの共通祖先の系譜を高速に探索することで、数十万人規模のゲノム情報に対する自然選択の解析を可能にしている。
(注6)理化学研究所2018年4月24日プレスリリース「全ゲノムシークエンス解析で日本人の適応進化を解明」
https://www.riken.jp/press/2018/20180424_2/index.html
(注7)大阪大学2020年1月20日プレスリリース「日本人と欧米人の適応進化に関わる遺伝子領域や形質を特定」
https://resou.osaka-u.ac.jp/ja/research/2020/20200120_1
(注8)理化学研究所2008年9月26日プレスリリース「1人あたり約14万個所のDNA 塩基多型を用いて日本人の集団構造を解明」
https://www.ims.riken.jp/pdf/20080926_1.pdf
(注9)東京大学医科学研究所2020年3月26日プレスリリース「大規模ゲノムの機械学習・次元削減手法により日本人集団の多様性を解明」
https://www.ims.u-tokyo.ac.jp/imsut/jp/about/press/page_00064.html
(注10)琉球大学 2014年9月16日プレスリリース:「ゲノム多様性データから明らかになった先史琉球列島人の移動」
https://www.u-ryukyu.ac.jp/wp-content/uploads/2021/01/press2014091602.pdf
(注11)琉球大学2021年1月21日プレスリリース「ゲノム解析によって明らかになった宮古諸島の人々の由来」
https://www.u-ryukyu.ac.jp/news/19465/
(注12)琉球大学2023年7月20日プレスリリース「全ゲノム配列解析により見えてきた沖縄島と宮古諸島の集団の形成過程」
https://www.u-ryukyu.ac.jp/news/47334/
(注13)沖縄バイオインフォメーションバンク:琉球大学では沖縄県民の健康・長寿増進を目指して、内閣府、文部科学省、沖縄県科学技術振興課の支援を受け、2016年に沖縄バイオインフォメーションバンクを立ち上げた。現在、このプロジェクトの一環として、ゲノムDNAなどの生体試料、ゲノム情報、臨床検査情報等の収集・解析を進めている。2023年8月末時点で25,000人以上の試料収集が完了している。
http://adgenome.skr.u-ryukyu.ac.jp/%e7%a0%94%e7%a9%b6%e5%86%85%e5%ae%b9/
https://amrc.skr.u-ryukyu.ac.jp/field/bio/
(注14)国立長寿医療研究センターバイオバンク:国内6つのナショナルセンター(国立高度専門医療研究センターの通称)でつくるバイオバンクネットワークの一員であり、高齢期に発症する認知症や骨関節疾患などを中心に、生体試料と情報を収集・保管し、国内外の医学研究に提供している。
https://www.ncgg.go.jp/ri/biobank/index.html
(注15)PCA(Principal Component Analysis):主成分分析と呼ばれる集団遺伝学で用いられる手法の一つ。PCAを用いてゲノム全体にわたる大量のSNP(一塩基多型)の情報を解析すると、各個人が遺伝的背景に応じた位置にプロットされるため、個人の間の遺伝的な距離を可視化することができる。 SNP(Single Nucleotide Polymorphism:一塩基多型)とは、ヒトゲノム上の個人差のひとつで、ヒトゲノムに約1000万か所存在する一塩基の違い。下図の黄色で強調した部分はSNPを表す。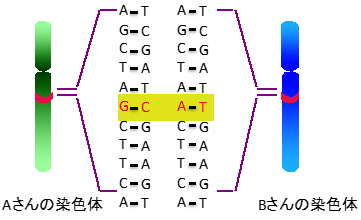
(注16)UMAP(Uniform Manifold Approximation and Projection):次元削減と呼ばれるデータ解析手法の一つ。 PCAと組み合わせることで、PCAだけでは区別できない詳細な個人間の遺伝的背景の違いを可視化することができる。
(注17)ハプロタイプ:特定のゲノム領域の近傍に位置する複数の一塩基多型等の個人差の組み合わせ。親から子へ遺伝情報が伝わる際には、一塊のハプロタイプ情報が受け継がれる。ハプロタイプが個人間で共有されている場合、その起原を祖先に遡ることができ、それを元に最近の集団史を推定することができる。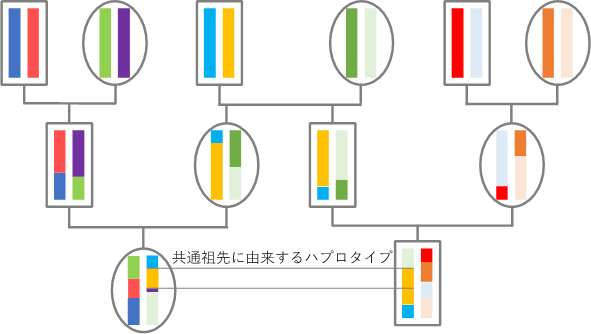
(注18) iHS(integrated haplotype score): ゲノム上の自然選択領域を検出する解析手法の一つ。自然選択上有利な対立遺伝子が含まれるハプロタイプは集団の中で、より長く保存され、その頻度が増加するという特徴を利用し、そのような領域をゲノム全体から探索することで自然選択領域を検出する手法。
(注19)Fast SMC:ゲノム上の自然選択領域を検出する手法の一つで、前述のASMC(注4)を拡張した遺伝統計解析手法。ASMCより高速で動作し、より最近の自然選択まで検出することができる。前述(注17)のiHSは現在のハプロタイプ情報、Fast SMCは過去の世代のハプロタイプ情報に基づいて自然選択を検出している。
(注20)IKZF2:IKAROS family zinc finger 2 :リンパ球の分化に関連し、IKZF2遺伝子の異常と後述(注25)の成人T細胞白血病・リンパ腫(ATL)の関連が報告されている。
(注21)HOXD:Homeobox D cluster:ホメオボックス遺伝子と呼ばれる転写因子を持つ遺伝子クラスターの一つ。Hox D1-13の遺伝子で構成され、受精卵から体が形成される発生や四肢の発生の過程で重要な役割を果たす。
(注22)CTNNA3:catenin alpha 3:ビンキュリン/αカテニンファミリーに属し、細胞間接着において重要な役割を果たす。CTNNA3の異常と不整脈原性右室心筋症との関連が報告されている。
(注23)MHC領域(Major Histocompatibility Complex;主要組織適合性複合体):ヒトの 6 番染色体短腕(6p21)上に存在し、その領域にある多重遺伝子族(遺伝子重複によって生じた遺伝子の中で相同性が高いもの)は、自己と非自己の認識、免疫応答の誘導に関与する白血球抗原をコードしている。HLA(human leukocyte antigen)領域とも呼ばれる。
(注24)対立遺伝子:ヒトは両親から1コピーずつ2つの相同染色体を受け継ぐ。それぞれの染色体上の同じ位置にあるSNPや遺伝子のことを対立遺伝子(アレル)という。対立遺伝子が同じ場合をホモ接合、異なる場合をヘテロ接合という。
(注25)HTLV-1(human T lymphotropic virus type 1: ヒトT細胞白血病ウイルスI型):一度感染すると生涯ウイルスを持ち続けるが、約95%の方は無症候性キャリアであり生涯関連疾患を発症しない。キャリアの一部の人は、成人T細胞白血病・リンパ腫(ATL)、HTLV-1関連脊髄症(HAM)、HTLV-1ぶどう膜炎(HU/HAU)などの関連疾患を発症する。
(注26)日中の過度の眠気や、通常起きている時間帯に自分では制御できない眠気が繰り返し起こることを特徴とする睡眠障害。
<謝辞>
本研究は文部科学省科研費・基盤研究(A)(寺尾知可史:20H00462)、新学術領域研究(研究領域提案型)(松波雅俊:18H05506)、日本医療研究開発機構(AMED)・難治疾患実用化研究事業(寺尾知可史:22ek0109555)、同ゲノム医療実現推進プラットフォーム・先端ゲノム研究開発(寺尾知可史:21tm0424220)、同革新的がん医療実用化研究事業(寺尾知可史:21ck0106642)、琉球大学・沖縄バイオインフォメーションバンクプロジェクト、沖縄・イノベーション・エコシステム共同研究推進事業(前田士郎)の支援を受けて実施されました。
<論文情報>
- 論文タイトル Natural selection signatures in the Hondo and Ryukyu Japanese sub-populations (本土集団と琉球列島集団の自然選択の痕跡)
- 雑誌名 Molecular Biology and Evolution
- 著者 Xiaoxi Liu#, Masatoshi Matsunami#, Momoko Horikoshi, Shuji Ito,
Yuki Ishikawa, Kunihiko Suzuki, Yukihide Momozawa, Shumpei Niida, Ryosuke Kimura, Kouichi Ozaki, Shiro Maeda, Minako Imamura*, Chikashi Terao*(# co-first authors, *co-corresponding authors) - DOI番号 10.1093/molbev/msad231
- アブストラクトURL https://academic.oup.com/mbe/article/40/10/msad231/7325950
