|
琉球大学大学院医学研究科の渡部匡史講師らと、京都薬科大学の共同研究チームによる研究成果が、スイスの学術雑誌「International Journal of Molecular Sciences」誌に掲載されました。 <発表のポイント>
|
<発表概要>
【研究の背景】
カポジ肉腫関連ヘルペスウイルス(KSHV: Kaposi’s sarcoma-associated herpesvirus)は、腫瘍やリンパ腫の原因となるがんウイルスです。初感染後に生涯にわたり潜伏感染を維持することが知られていますが、通常の健康状態であればウイルスによる病気は生じないケースがほとんどです。しかし、沖縄県宮古島ではこのウイルスによる腫瘍、古典型カポジ肉腫(注2)の発生率が高いことが報告されています。
KSHVの遺伝情報はDNAによって保持されています。ウイルスが増えるときには、ウイルスDNAを鋳型として、中間体であるRNAを介して、最終的にウイルスタンパク質をつくります。ヘルペスウイルスのウイルスタンパク質は、自身の増殖に有利なように感染した細胞内の環境を変えてしまうもの、自身のDNAを複製するもの、ウイルス粒子のパーツとなるものなど、多種多様です。
ヘルペスウイルスは、潜伏感染時には宿主細胞に備わっているタンパク質群を利用して、ウイルスタンパク質の設計図となるメッセンジャーRNAをつくり、自己保存をはかります。
この際には、複数の宿主タンパク質から構成される転写開始前複合体(注3)が利用されます。
KSHVと一部のヘルペスウイルスは、潜伏感染状態から盛んにウイルスをつくる状態に移行したときに、宿主の転写開始前複合体を利用せずにメッセンジャーRNAをつくり始める場合もあることが明らかになってきました。この際にはウイルスタンパク質群から構成されるウイルス性転写開始前複合体(注4)を利用することが、分かってきました。
ウイルス性転写開始前複合体は、少なくとも6種類のウイルスタンパク質から構成されていると考えられていますが、ウイルスタンパク質どうしの結合のしかたは不明な部分が多くあります。その大きな理由の一つとして、各ウイルスタンパク質の構造がわからないことがあげられます。
【研究の内容】
渡部らは、2つのウイルスタンパク質(KSHV ORF18とKSHV ORF30)間の結合度合いについて、定量的に調べるために二分子蛍光補完(BiFC)法を用いました。蛍光タンパク質を2分割したタンパク質断片を、2つのウイルスタンパク質それぞれに融合させたタンパク質を培養細胞内で発現させ、細胞内の蛍光タンパク質を計測しました。さらに結合領域や結合アミノ酸残基を絞り込んでいったKSHV ORF18変異体を評価したところ、ORF18上の4アミノ酸が重要であることが分かりました。
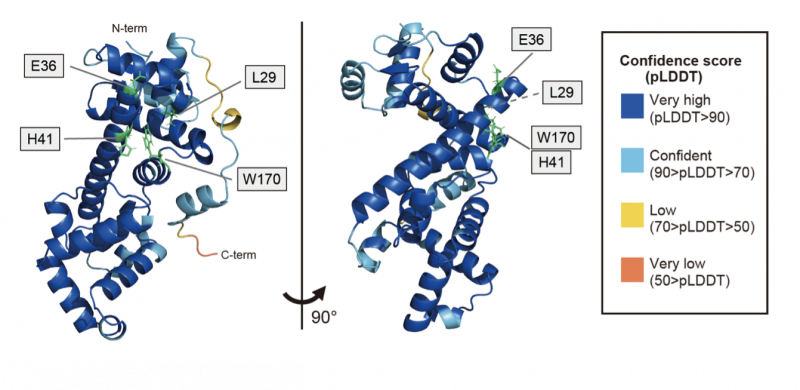
図1:AlphaFold2によるKSHV ORF18構造予測モデルと結合に重要なアミノ酸残基
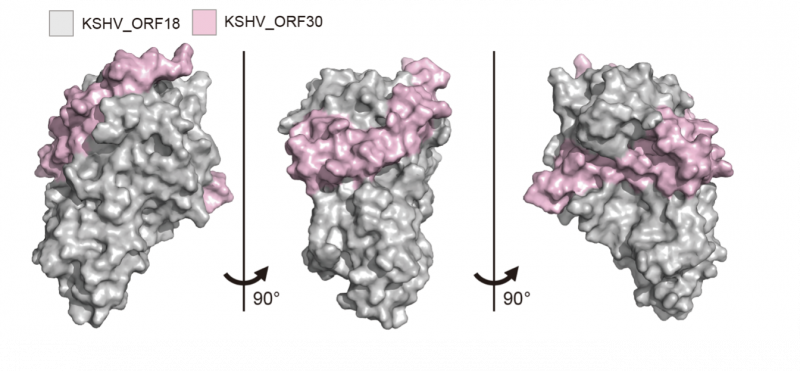
図2:AlphaFold2によるKSHV ORF18-ORF30の複合体構造予測モデル
これらのアミノ酸がタンパク質間の結合にどう関わっているのかを明らかにするためには、タンパク質構造情報が必要です。しかし、今回着目した2つのウイルスタンパク質(KSHV ORF18とKSHV ORF30)の3次元構造は、実験的に証明されてい ません。そこで、昨年2021年にDeepmind社から公開された、アミノ酸配列情報からタンパク質3次元構造を予測する人工知能(AI)アルゴリズムAlphaFold2(注5)を利用しました。Deepmind社は囲碁AIプログラムAlphaGoを開発したことで広く知られています。AlphaFold2の予測精度は、既存のプログラムよりも遥かに優れており、今後の生命科学分野に大きなイノベーションをもたらしうると考えられています。
AlphaFold2により、KSHV ORF18のアミノ酸配列情報から確度の高い構造モデルが得られ(図1)、さらにKSHV ORF30との結合モデルも得ることができました(図2)。これらのモデルと照らし合わせると、結合に重要なKSHV ORF18上の4アミノ酸は、いずれもKSHV ORF30との結合面やその近い位置に存在していることが示されました(図3)。このことは、実験データとAI予測構造モデルが互いに結果を支持しあっていることを示しており、本研究の結果得られた結合予測モデルは強く支持されうるものだと考えられます。
【今後の展望】
カポジ肉腫関連ヘルペスウイルスそのものに対して有効な薬剤は現時点でありません。ウイルス性転写開始前複合体は、ウイルスが作り出されるのに必須な複合体です。この複合体が形成されるのを阻止するような物質を発見すれば、カポジ肉腫治療に大きな進歩をもたらすことになると考えられます。その足場づくりとして、本研究で実施したような結合メカニズムの解明は必要不可欠です。
また、同じヘルペスウイルスのヒトサイトメガロウイルスやEBウイルスもそれぞれ同様のウイルス性転写開始前複合体を持つことから、これらの創薬にもつながる可能性があります。
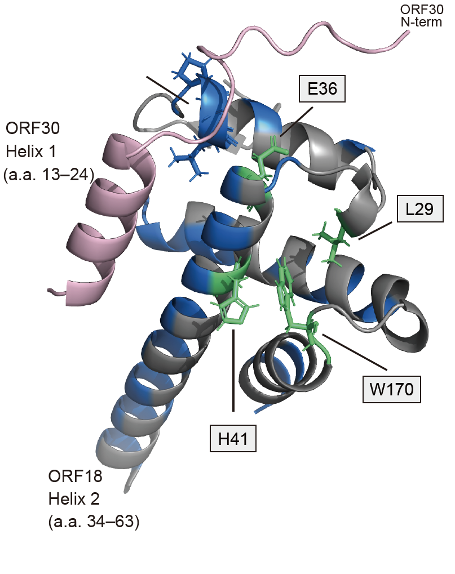
図3:KSHV ORF18-ORF30の結合予測領域(拡大)
<用語解説>
(注1)カポジ肉腫関連ヘルペスウイルス:カポジ肉腫から発見されたヘルペスウイルス。カポジ肉腫以外にもB細胞系リンパ腫の原因ともなる。アフリカ諸国では感染・発症率ともに高く臨床的に問題となっている。
(注2)古典型カポジ肉腫:カポジ肉腫関連ヘルペスウイルス感染のみを原因とする上皮系がん。表皮上に隆起した紫斑が形成されることが多い。地中海沿岸の高齢男性での発症例が多く1800年代後半にモーリッツ・カポジにより報告された。一般的に日本全体で発症率の高いカポジ肉腫は、HIV感染を伴ったAIDS関連カポジ肉腫が多数を占める。
(注3)転写開始前複合体:TATAボックス結合タンパク質、基本転写開始因子群から構成される複合体、RNAポリメラーゼIIをゲノム上の転写開始領域にリクルートして、メッセンジャーRNAの合成を開始する。
(注4)ウイルス性転写開始前複合体:カポジ肉腫関連ヘルペスウイルスのウイルス性タンパク質から構成される複合体。宿主の転写開始前複合体と同様にRNAポリメラーゼIIのリクルートに関わる。ウイルス膜タンパク質などのウイルス粒子を構成するタンパク質の発現調節に深く関わっている。
(注5)J. Jumper, R. Evans, A. Pritzel, T. Green, M. Figurnov, O. Ronneberger, et al., Highly accurate protein structure prediction with AlphaFold, Nature , 596, 583-589, 2021
<研究支援体制>
本研究は、文部科学省科研費21K08509, 18K14910(研究代表:渡部匡史)、18K06642(研究代表:藤室雅弘(京都薬科大学))、琉球大学若手研究者支援研究費20SP04102(研究代表:渡部匡史)、琉球大学医学部先端医学研究支援事業(研究代表:渡部匡史)の支援を受けて行われました。また、沖縄県委託事業「令和2年度健康・医療産業における情報技術活用促進事業:バイオインフォマティシャン人材育成講座」の受講による学術的支援を受けました。
<論文情報>
(1)タイトル:Biomolecular Fluorescence Complementation Profiling and Artificial Intelligence Structure Prediction of the Kaposi’s Sarcoma-Associated Herpesvirus ORF18 and ORF30 Interaction
(和訳)二分子蛍光補完法とAIタンパク質構造予測を用いたカポジ肉腫関連ヘルペスウイルスORF18とORF30間の相互作用解析
(2)雑誌名:International Journal of Molecular Sciences
(3)著者:Yoshiko Maeda†,Tadashi Watanabe†, Taisuke Izumi, Kazushi Kuriyama, Shinji Ohno and Masahiro Fujimuro* (†; Equal contribution, *; Corresponding author)
(4)DOI:https://doi.org/10.3390/ijms23179647
(5)URL:https://www.mdpi.com/1422-0067/23/17/9647